Los polimorfismos de un único nucleótido (Single Nucleotide Polimorphism=SNP) son una de las variaciones más estudiadas en todos los genomas y, especialmente, en humanos. Estos SNPs (se lee esnips) no son más que variaciones de una sola base de la secuencia de ADN. Ahora también se consideran SNPs a unos cuantos nucleótidos que varían, como también pequeñas inserciones y deleciones pueden ser consideradas como SNP. 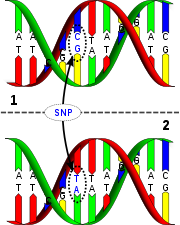
Es una de las tareas en las que estoy embarcado en estos momentos y con el avance de las tecnologías cada vez se conoce más y se tienen más datos sobre este tipo de variaciones. Sólo un dato: el 90% de todas las variaciones del genoma humano pertenecen a SNPs. Por lo tanto, son numerosos los estudios que se basan en estos SNPs para detectar enfermedades. Y no sólo en la especie humana.
Quería compartir una base de datos muy importante en este campo: la SNPedia. Consta de un catálogo de los SNPs que se van descubriendo y secuenciando. Incluso están clasificándolos por genomas utilizados, genes involucrados y también medicinas que actúan mejor o peor dependiendo de tener o no estas variaciones.
Os dejo este enlace para la SNPedia y otro para su blog, donde actualmente han publicado los 10 SNPs más relevantes del pasado año. Me encantan estas cosas.

2 comments