Las fotos del ADN
Ahora que estoy terminando de escribir la ansiada tesis doctoral, me hubiera gustado poder enseñar como he ido trabajando en el laboratorio día a día. Una de las labores cotidianas es realizar fotografías de los geles de electroforesis que contienen los fragmentos de ADN previamente amplificados mediante PCR para poder analizarlos posteriormente con más detenimiento. Esas imágenes se han podido ver en infinidad de documentales y series de televisión. Son esas fotografías en las que salen una serie de bandas que pertenecen a cada uno de los productos de PCR (aunque también pueden ser digestiones con enzimas u otro tipo de análisis a nivel molecular).
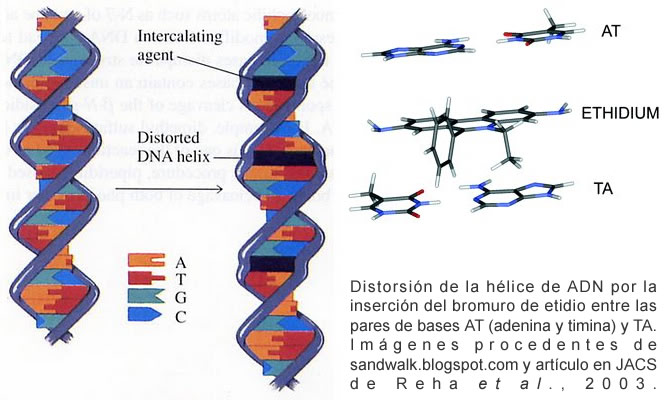
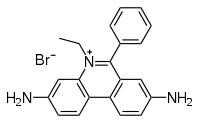
Resumiré las nociones básicas de por qué se ven las bandas de ADN gracias al bromuro de etidio (cada día en más desuso por la utilización de otros agentes intercalantes como el Sybr green, con relativo menor peligro mutagénico) y el por qué de su «iluminación» al incidir los rayos ultravioleta. Cuando se expone el bromuro de etidio (ó Bromuro de 3,8-diamino-5-etil-6-fenilfenantridinio) a luz ultravioleta, emite una luz roja-anaranjada, que se intensifica unas 20 veces después de haberse unido a una cadena de ADN. Este efecto es debido al aumento de la hidrofobia del medio. Como el bromuro de etidio se intercala en el ADN, esta sustancia tiene un poderoso efecto mutágenico y, posiblemente puede ser cancerígeno o teratógeno.
Desde que lanzaron el conocido Sybr green los laboratorios Invitrogen®, parecía que el miedo que siempre nos metían a todos los científicos sobre lo malo que era este compuesto desaparecería con el nuevo «invento verde». Esta molécula se introduce en la estructura secundaria de la doble hélice del ADN y se acopla energéticamente a los ácidos nucleicos que lo forman, de manera que se incrementa notablemente su tasa de emisión fluorescente. Este fenómeno se conoce como transferencia de energía mediante resonancia de fluorescencia. El complejo resultante ADN-SYBR Green presenta el pico de absorción en λ = 498 nm y el pico de emisión en λ = 522 nm (correspondiente a la zona verde del espectro, de ahí su nombre). Ese «miedo» sobre la genotoxicidad del bromuro de etidio que nos intercalaban en nuestras mentes no está muy bien determinado. Como se explicó en 2006 en rrresearch.fieldofscience.com por Rosie Redfield, esa posibilidad mutagénica no está demostrada y las cantidades para que ocurra son increíblemente elevadas. Casi todas las marcas que lo distribuyen muestran los resultados del test de Ames que demuestran el pase de este Sybr green como no mutagénico. Sin embargo, parece ser que existen estudios (ver artículo anteriormente enlazado) en los que se demuestra una mayor toxicidad en ratones con este compuesto que con el bromuro de etidio. Lo malo es que no se suelen explicar todos los pros y contras de cada compuesto como se debería. Y los agentes de ventas de las casas que comercializan estos compuestos tampoco conocen todo sobre ellos. Y no tienen por qué ser completamente transparentes si omitiendo datos consiguen ventas. Lo importante no es echar culpas si no ser precavidos e informarse debidamente.
Una vez dicho todo esto sobre lo que esconden nuestros geles teñidos, quería mostrar que los investigadores del centro TNAU Genomics & Proteomics parece que me han leído el pensamiento y han elaborado un vídeo sobre esta toma de imágenes. Lo bueno es que es exactamente el mismo equipo que usamos en nuestros laboratorios. Tanto el transiluminador Gel Doc como el software Quantity One de BioRad®. El vídeo está en un inglés con acento hindú, pero muy fácil de seguir. Sin más dilación, os dejo con el documento multimedia:
[youtube]http://www.youtube.com/watch?v=a38-lYuPhkQ[/youtube]
Este es uno de los equipos. Para los geles de poliacrilamida de toda la vida podemos tomar las fotos con un densitómetro (como un escáner) y luego están los equipos automatizados de electroforesis capilar que te suministran los datos directamente en formato digital para analizarlos tranquilamente. Eso puede ser tema para otro artículo.
Y vosotros ¿qué compuestos y aparatos utilizáis para estas «tareas cotidianas»?
Este post participa en la VI Edición del Carnaval de Biología, , que organiza @Copepodo en su blog Diario de un Copépodo y también en el VIII Carnaval de Química organizado por @lualnu10 en su blog “Caja de Ciencia”.


Escuchando: El golpeo de las teclas en el portátil ;-P
Continue reading