Amplificación del ADN: la PCR
La amplificación del ADN mediante PCR no podía faltar en este blog. Ya he podido comentar como se trabaja con el material hereditario anteriormente. Mejor dicho, cómo se prepara para poder sacarle partido. Para poder tener varias copias de una región específica de ADN, se utiliza la tecnología de la Reacción en Cadena de la Polimerasa (Polimerase Chain Reaction = PCR).
Comienzos de la PCR
Este «gran invento» de la PCR se lo debemos al premiado Nobel en Química de 1993 Kary B. Mullis. Se le ocurrió realizar in vitro las condiciones necesarias para conseguir copias de fragmentos de ADN.
El ADN está en forma de doble hélice. Para que se pueda amplificar cada hebra, es necesario que se rompan los enlaces existentes para que se mantenga esa estructura. La idea consiste en reproducir ese vaivén molecular en un tubo de ensayo. Y así pudo hacerlo con la ayuda de todos los compañeros de la empresa en la que Kary B. Mullis trabajaba: Cetus Corporation.
Según nos puede contar la historia cercana de la Biología Molecular, el señor Mullis tuvo la brillante idea mientras conducía por las carreteras californianas a horas nocturnas. Observó el ir y venir de los coches cruzándose por las diferentes vías. De pronto, paró el coche y comenzó a pensar sobre el proceso de amplificación del ADN in vitro y como con tan sólo 20 ciclos podrían obtenerse la friolera de un millón de moléculas a partir de dos hebras de dicho ácido desoxirribonucleico.
Cuando volvió al trabajo lo puso en práctica. Funcionaba. La sencillez de todo el proceso hizo que hubiera muchos desconfiados dentro del ámbito de la Genética molecular. Poco tiempo después, otro premio nobel llamado Joshua Lederberg se quedó perplejo mirando el póster explicativo de Mullis con todo el proceso detallado. Le preguntó «¿funciona?», a lo que después espetó un «¿Por qué no se me habría ocurrido a mí?».
Amplificación del ADN paso a paso
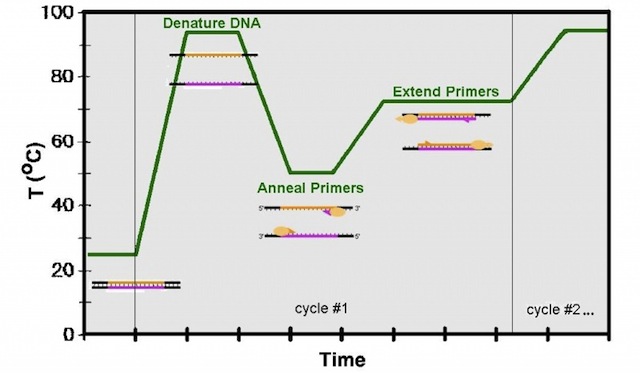
Este proceso se realiza elevando la temperatura aproximadamente a 95 ºC durante un breve período de tiempo. Se denomina desnaturalización.
Posteriormente se necesita que los cebadores (oligonúcleótidos o secuencias cortas de ADN de unos 20 nucleótidos diseñadas para que flanqueen una zona específica de ADN que se quiera amplificar) se anclen a sus secuencias complementarias. L
a temperatura juega un papel importantísimo, puesto que cada pareja de cebadores (siempre se habla de parejas puesto que se debe tener un primer o cebador por un lado y otro por el otro para que se amplifique el mismo fragmento por ambos lados y producir la amplificación de la zona flanqueada) hibrida (se une al ADN) a una temperatura que, generalmente, puede variar entre 45 y 65 ºC. Aunque hay protocolos en los que se utilizan variaciones de temperatura para obtener un mejor rendimiento, pero esto lo comentaré en artículos posteriores.
Finalmente, se necesita una extensión de los fragmentos flanqueados por los primers gracias a la acción de una molécula llamada Polimerasa y que tiene su temperatura óptima de reacción a 72 ºC (aunque se utiliza también una temperatura óptima de 68 ºC, dependiendo de la casa que suministre la polimerasa).
Resumen de la PCR
Por lo tanto y en resumen se tiene en todo el proceso 3 fases:
- Desnaturalización.
- Hibridación de los cebadores.
- Extensión de los fragmentos. Al final de todo el programa, se introduce una fase de extensión más larga para que se termine de obtener un mayor número de copias.
Todo este proceso se realiza en los termocicladores: las reacciones se preparan en frío y los tubos o placas de reacción se depositan en estos aparatos que son programados para realizar los ciclos que he explicado antes. Un esquema de programa es el que pongo en la imagen siguiente:
En cada ciclo de amplificación del ADN, el fragmento diana aumentará en el número de copias de forma exponencial. Esto provoca que, al final de un programa básico, se obtengan aproximadamente hasta 100 millones de copias del fragmento deseado.
Consideraciones sobre la amplificación del ADN por PCR
Las variaciones de tiempo dependen principalmente de la longitud de los fragmentos. Cuanto más tiempo mayor es el fragmento a amplificar. La clave de una buena eficiencia depende de los diseños de las reacciones de PCR que se deben ajustar a las condiciones de cada reacción.
Al principio, las polimerasas que se utilizaban no eran termorresitentes. Esto provocaba que, en cada ciclo, había que añadir polimerasa para que se pudiera extender la amplificación del ADN. Ahora se utilizan polimerasas que permiten ser añadidas en la preparación de las reacciones y se puede olvidar de ello.
No más importante, cuidado con el material a ser utilizado: el calibrado de las micropipetas es esencial y la «limpieza» del resto de materiales y lugar de trabajo pueden ser clave.
Me gustaría explicarlo todo: como se diseñan los cebadores, la realización de la reacción de PCR y todos los componentes, los estudios que se pueden derivar…etc, pero igual no acabo con el artículo y creo que la base sí que la he plasmado.
Una cosa es clara: sin la propia evolución en los conocimientos sobre el ADN y su comportamiento no se podría haber llegado a la situación de Mullis. Personas como James D. Watson, Francis Crick, Rosalind Franklin, Arthur Kornberg, H. Gobind Khorana o Thomas D. Brock tienen en sus espaldas haber sembrado poco a poco la información que ayudó a Kary B. Mullis a desarrollar su idea (que puede tener cierta controversia con el trabajo presentado por Kjell Kleppe).
Información adicional sobre la amplificación del ADN por PCR
Lo mejor es partir del propio escrito original de Kary B. Mullis. Después de generar las correspondientes patentes se puede encontrar por la red un par de documentos datados en 1986 y 1987 respectivamente con los títulos «Specific enzymatic amplification of DNA in vitro: the polymerase chain reaction» y «Specific Synthesis of DNA in vitro via a Polymerase-Catalyzed Chain Reaction».
Otra lectura que recomiendo encarecidamente es mi monográfico dedicado a la PCR que escribí en su momento para Journal of Feelsynapsis. Como la revista se ha reconvertido en Principia, cualquiera que desee leerlo puede pedirme una copia que se la enviaré con honores.
Este vídeo explica todo bastante bien con un inglés muy fácil de seguir. Incluso se observan los fragmentos que no son específicos (que no se buscan amplificar pero que se obtienen de todas formas por la hibridación de los primers)
[youtube]http://www.youtube.com/watch?v=eEcy9k_KsDI[/youtube]
Referencia del vídeo: Essential Cell Biology, 3rd Edition. Alberts, Bray, Hopkin, Johnson, Lewis, Raff, Roberts, & Walter
Si se tuviera alguna duda al respecto, los comentarios e email están a disposición de cualquiera (y abiertos las 24 horas del día los 365 días del año).
Escuchando: el sonido de las campanas de Papá Noël…JOJOJO!!
Continue reading